
Profesor Titular B T.C.
SNI Nivel II
Ubicación: Laboratorio 103. Edificio E
Facultad de Química
Tel: +52 (55) 56225277
Correo institucional: cesy@unam.mx
Correo alterno: cesyd@yahoo.com ![]() Presentación de Grupo de Investigación (TDD)
Presentación de Grupo de Investigación (TDD)
Formación Académica
Lic. Bioquímica, Universidad de la Habana
Ph.D. Bioquímica, UNAM
Postdoc LSUHSC USA
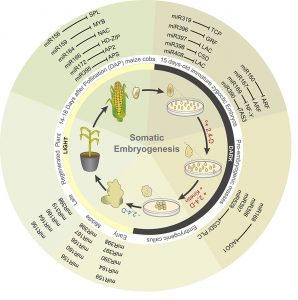
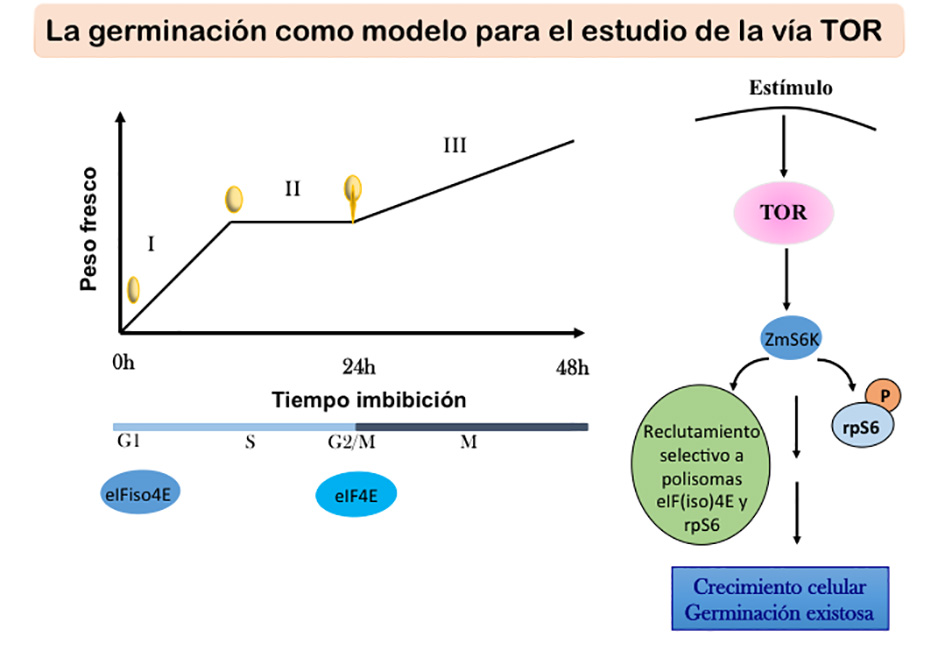
Líneas de Investigación Regulación de la traducción mediada por diferentes miembros de la familia eIF4E durante el desarrollo de plantas y en respuesta a estrés Los factores de inicio de la traducción eIF4E, eIF(iso)4E y nCBP en plantas pertenecen a la familia de proteínas tipo eIF4E que unen a la estructura 7mGpppN presente en el extremo 5’ de los mRNA. Mediante su interacción con otras proteínas y la unión al 5’cap, estos factores permiten regular el inicio de la traducción de los mRNAs celulares dependiendo de las condiciones y etapa del desarrollo. En nuestro laboratorio, estamos interesados en abordar la función de eIF4E, eIF(iso)4E y nCBP en la traducción selectiva de mRNAs en respuesta a señales de desarrollo y presencia de estrés biótico y abiótico. Hemos encontrado que los complejos proteicos formados por estas proteínas son diferentes durante la germinación de maíz y promueven traducción selectiva de mRNAs. Por otro lado, en el modelo de Arabidopsis thaliana, abordamos su función en la respuesta a estrés por temperatura (frío o calor) mediante el análisis de mutantes simples y plantas sobreexpresoras de algunas de las isoformas. Regulación de la expresión genética por RNAs pequeños durante la embriogénesis somática de maíz La embriogénesis somática permite la propagación en masa de plantas, la producción de semillas sintéticas, la transformación genética de algunas especies y el análisis de la expresión genética en este proceso de desarrollo alternativo de las plantas. Los mecanismos de regulación mediados por microRNAs (miRNAs) y pequeños RNA de interferencia (siRNA) son relevantes para el silenciamiento de genes y las modificaciones epigenéticas en respuesta a estímulos, así como para la estabilidad y traducción de RNAs mensajeros a nivel post-transcripcional. Esto puede tener un papel fundamental en la re-programación genética que implica la inducción y mantenimiento de los cultivos de células indiferenciadas (callos o suspensiones celulares) y para la posterior regeneración de plantas. El objetivo principal de este proyecto es identificar a RNAs pequeños que modifican su acumulación a lo largo del proceso de Embriogénesis Somática y analizar su papel en la regulación de mRNAs diana que participan en las respuestas a señales de desarrollo, hormonales y de estrés para garantizar el éxito de la regeneración de plantas. Participación de la vía de transducción de señales mediada por Target of Rapamycin (TOR) en la regulación traduccional durante la germinación y respuesta a estrés en maíz TOR es una cinasa que representa el sensor integrador de señales de metabolismo en los eucariontes. En animales, una de las múltiples conexiones de TOR para integrar la respuesta celular a condiciones ambientales es la regulación de la traducción de mRNAs a nivel global y específico. En plantas, no se ha podido establecer el mecanismo mediante el cual TOR impacta sobre la traducción, aunque sí se conoce su conexión con otros sensores conservados del metabolismo celular. En este proyecto estamos investigando la relación entre la actividad de TOR, los complejos de unión a 5’cap mediados por eIF4E y eIF(iso)4E y la síntesis de proteínas durante la germinación de maíz, en respuesta a auxinas y estrés por calor. Publicaciones: Herrera-Díaz, J., Jelezova, M.K., Cruz-García, F., Dinkova, T.D. (2018) Protein Disulfide Isomerase (PDI1-1) differential expression and modification in Mexican malting barley cultivars. PLoS ONE 13(11): e0206470. https://doi.org/10.1371/journal Alejandri-Ramírez, N.D., Chávez-Hernández, E.C., Contreras-Guerra, J.L., Reyes, J.L., Dinkova, T.D. (2018) Small RNA differential expression and regulation in Tuxpeño maize embryogenic callus induction and establishment. Plant Physiology and Biochemistry 122: 78-89. https://doi.org/10.1016/j.plaphy.2017.11.013 ISSN: 0981-9428. Ochoa-Olmos, O.E., León-Domínguez, J.A., Contreras-Torres, F.F., Sanchez-Nieto, S., Basiuk, E.V., Dinkova, T.D. (2016) Transformation of Plant Cell Suspension Cultures with Amine-functionalized Multi-Walled Carbon Nanotubes. J. Nanosci. Nanotechnol. 16: 7461-7477. Dinkova, T.D., Martinez-Castilla, L., Cruz-Espindola, M.A. (2016) The Diversification of eIF4E Family Members in Plants and Their Role in the Plant-Virus Interaction. In: Evolution of the protein synthesis machinery and its regulation (Eds: Jagus, R., Hernandez, G.) Springer International Publishing, Switzerland 2016, pp. 187-205. ISBN 978-3-319-39467-1. Chávez-Hernández, C., Alejandri-Ramírez, N.A., Juárez-González, V.T., Dinkova, T.D. (2015) Maize miRNA and target regulation in response to hormone depletion and light exposure during somatic embryogenesis. Frontiers in Plant Science 6: 555; doi: 10.3389/fpls.2015.00555; ISSN: 1664-462X Contreras-Paredes, C.A., Silva-Rosales, L., Daròs, J.-A., Alejandri-Ramírez, N.D., Dinkova, T.D. (2013) The absence of eukaryotic initiation factor eIF(iso)4E affects the systemic spread of a tobacco etch virus isolate in Arabidopsis thaliana. Molecular Plant-Microbe Interaction 26: 461-470. Lázaro-Mixteco, P.E., Nieto-Sotelo, J., Swatek, K.N., Houston, N.L., Mendoza, G., Thelen, J.J., Dinkova, T.D. (2012) The absence of Heat Shock Protein Hsp101 affects the proteome of the mature and germinating maize embryo. Journal of Proteome Research 11: 3246-3258. dx.doi.org/10.1021/pr3000046 Martínez-Silva, A.V., Aguirre-Martínez, C., Flores-Tinoco, C.E., Alejandri-Ramirez, N.D., Dinkova, T.D. (2012) Translation initiation factor AteIF(iso)4E is involved in selective mRNA translation in Arabidopsis thaliana seedlings. PLoS ONE 7(2): e31606. doi:10.1371/journal.pone.0031606 Proyectos: MECANISMOS DE REGULACIÓN mRNA-ESPECÍFICOS MEDIADOS POR MIEMBROS DE LA FAMILIA EIF4E EN PLANTAS (CONACYT 81708 Octubre 2008 – Septiembre 2011) PAPEL DE LAS PROTEÍNAS QUE INTERACCIONAN CON LOS COMPLEJOS DE UNIÓN A CAP (EIF4F/EIFISO4F) EN LA REGULACIÓN TRADUCCIONAL EN MAÍZ (PAPIIT IN204309 Enero 2009 – Diciembre 2011) PAPEL DE LOS MICRO RNAS (MIRNAS) EN LA REGULACIÓN DE LA EXPRESIÓN GENETICA DURANTE LA EMBRIOGENESIS SOMATICA DE MAÍZ (PAIP 6290-05) Identificación de miRNAs expresados diferencialmente durante la inducción de tejido indiferenciado (callos embriogenicos) y mantenimiento del callo (division celular) y regeneracion de plantulas (diferenciación) Grupo de Investigación:Desarrollo Profesional
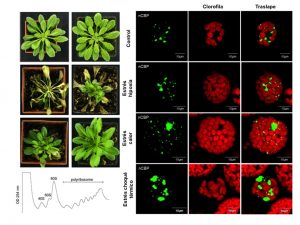
http://dx.doi.org/10.1094/MPMI-09-12-0225-R
 Vasti Thamara Juárez González (doctorado) Proyecto: “Papel de las vías de silenciamiento mediadas por sRNAs durante la inducción de callos embriogénicos de maíz”
Vasti Thamara Juárez González (doctorado) Proyecto: “Papel de las vías de silenciamiento mediadas por sRNAs durante la inducción de callos embriogénicos de maíz” Kenia Salazar Díaz (maestría) Proyecto: “Papel de los factores de inicio de la traducción eIF4E y eIF(iso)4E durante el estrés por frío en Arabidopsis thaliana”
Kenia Salazar Díaz (maestría) Proyecto: “Papel de los factores de inicio de la traducción eIF4E y eIF(iso)4E durante el estrés por frío en Arabidopsis thaliana” Brenda Nieto Rivera (maestría) Proyecto: “Edición de genes correspondientes a la familia eIF4E en Arabidopsis thaliana mediante CRISPR-Cas”
Brenda Nieto Rivera (maestría) Proyecto: “Edición de genes correspondientes a la familia eIF4E en Arabidopsis thaliana mediante CRISPR-Cas” Marlon Adrián Pulido Torres (maestría) Proyecto: “Respuestas mediadas por miembros de la familia eIF4E al estrés abiótico en plantas”
Marlon Adrián Pulido Torres (maestría) Proyecto: “Respuestas mediadas por miembros de la familia eIF4E al estrés abiótico en plantas” Grecia María Olea Badillo (licenciatura) “Expresión de componentes de la vía TOR durante la germinación de maíz”
Grecia María Olea Badillo (licenciatura) “Expresión de componentes de la vía TOR durante la germinación de maíz” Brenda Anabel López Ruiz (doctorado) Proyecto: “Función de los tasi-RNAs durante la regeneración de plantas a partir de callos embriogénicos de maíz”
Brenda Anabel López Ruiz (doctorado) Proyecto: “Función de los tasi-RNAs durante la regeneración de plantas a partir de callos embriogénicos de maíz” Eduardo Luján Soto (doctorado) Proyecto: “Estudio de la regulación transcripcional de los genes MIR528 en maíz (Zea mays)”
Eduardo Luján Soto (doctorado) Proyecto: “Estudio de la regulación transcripcional de los genes MIR528 en maíz (Zea mays)” Mayra Aquino Luna (maestría) Proyecto: “Regulación de miembros de la familia ARF por sRNAs durante la embriogénesis somática de maíz”
Mayra Aquino Luna (maestría) Proyecto: “Regulación de miembros de la familia ARF por sRNAs durante la embriogénesis somática de maíz” Karen Guadalupe Calderón Valencia (licenciatura) “Efecto de auxinas y estrés por calor en la vía TOR durante la germinación y establecimiento de plántulas de maíz”
Karen Guadalupe Calderón Valencia (licenciatura) “Efecto de auxinas y estrés por calor en la vía TOR durante la germinación y establecimiento de plántulas de maíz”